liuyujie0136's Website

A website for self learning, collecting and sharing.
使用JCVI进行MCScan共线性(synteny)分析与可视化
https://github.com/tanghaibao/jcvi
https://sr-c.github.io/2019/01/11/jcvi-MCscan/
https://blog.csdn.net/u012110870/article/details/102804593
安装
推荐使用conda安装jcvi,并安装依赖软件BEDTools、EMBOSS和LAST
conda create --name jcvi python=3.9 jcvi bedtools emboss last
在此之前,建议按照官网教程安装texlive,而不要使用conda中的texlive-core
wget https://mirror.ctan.org/systems/texlive/tlnet/install-tl-unx.tar.gz
tar -xzf install-tl-unx.tar.gz
cd install-tl-*
perl install-tl
# 若无管理员权限需要安装在家目录
# 在交互界面按"D",再按"1"
# 更改"TEXDIR"至合适位置
# 再按提示完成安装(约需1.5h)
准备数据
jcvi支持直接从phytozome下载数据(需要先注册)
查看可下载的物种,常见的植物物种都可以直接下载
python -m jcvi.apps.fetch phytozome
下载与整理数据
# download sequences and coordinates of grape and peach
python -m jcvi.apps.fetch phytozome Vvinifera,Ppersica
# convert the GFF to BED file
python -m jcvi.formats.gff bed --type=mRNA --key=ID --primary_only Vvinifera_145_Genoscope.12X.gene.gff3.gz -o grape.bed
python -m jcvi.formats.gff bed --type=mRNA --key=ID --primary_only Ppersica_298_v2.1.gene.gff3.gz -o peach.bed
# clean headers to remove description fields from Phytozome FASTA files
python -m jcvi.formats.fasta format Vvinifera_145_Genoscope.12X.cds.fa.gz grape.cds
python -m jcvi.formats.fasta format Ppersica_298_v2.1.cds.fa.gz peach.cds
共线性分析
python -m jcvi.compara.catalog ortholog --no_strip_names grape peach
程序默认按照可用线程,调用全部进行LAST比对
可视化
点阵图
在共线性分析后,程序会自动绘制点阵图。手动运行方式如下
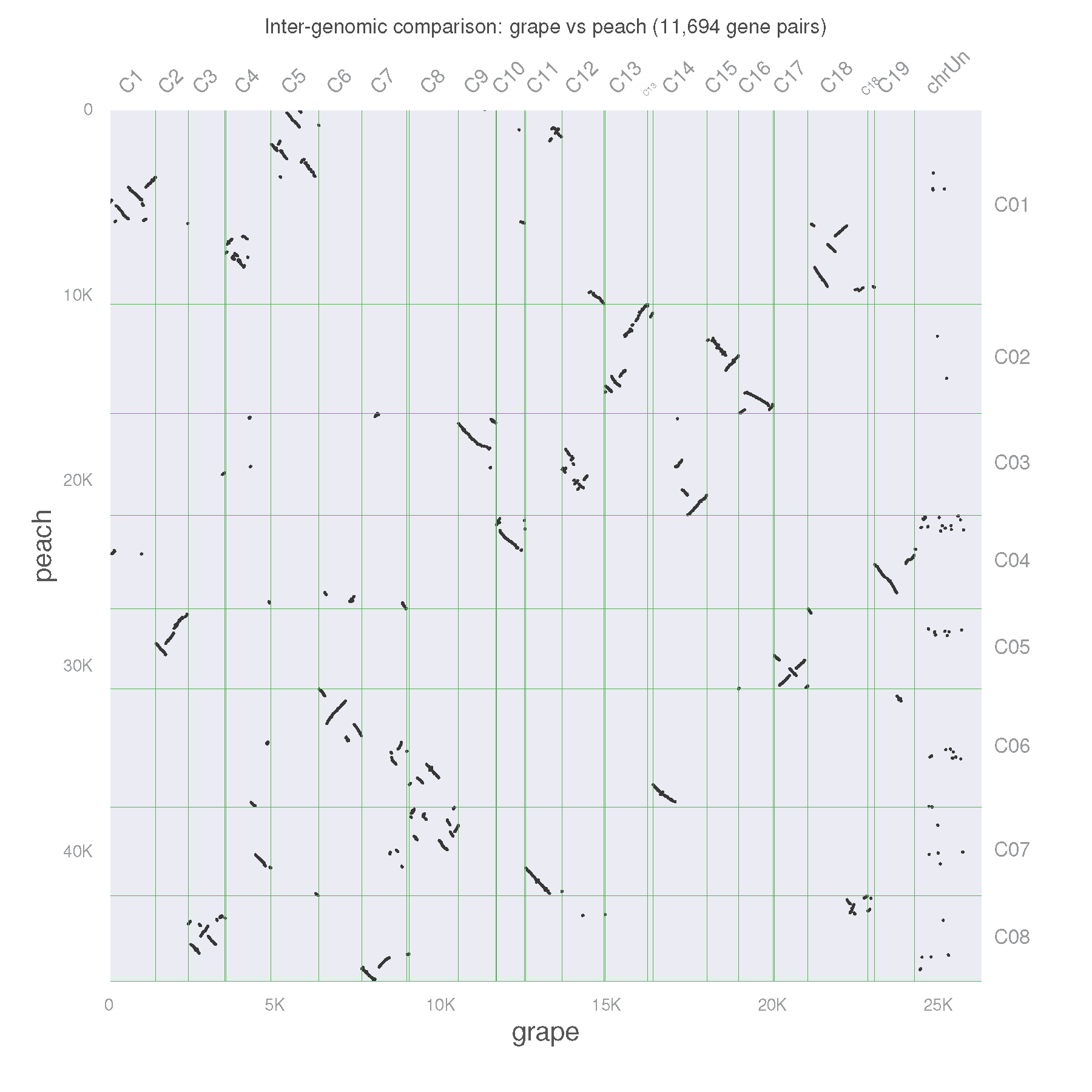
python -m jcvi.graphics.dotplot grape.peach.anchors
在两者的点阵图中,不论水平方向或者竖直方向查看,都可以发现不超过3次的共线性区域。这说明,🍇和🍑之间发生了基因组三倍体化的事件,使得出现了这样3:3的模式。
如果仔细观察,还可以发现3个共线性区域中常常有一个信号更强,对应着两个基因组之间的直系同源区域。如果我们只想要得到这些1:1直系同源的区域呢?我们只需要重复之前的比对,同时加上选项--cscore=.99即可。C-score是由LAST比对区域到BLAST比对区域的比值确定。(C-score is defined by the ratio of LAST hit to the best BLAST hits to either the query and hit)。0.99的C-score阈值有效地过滤LAST比对结果,从而得到最佳相互比对结果(reciprocal best hit, RBH)。
# remove old file
rm grape.peach.last.filtered grape.peach.anchors
python -m jcvi.compara.catalog ortholog grape peach --cscore=.99
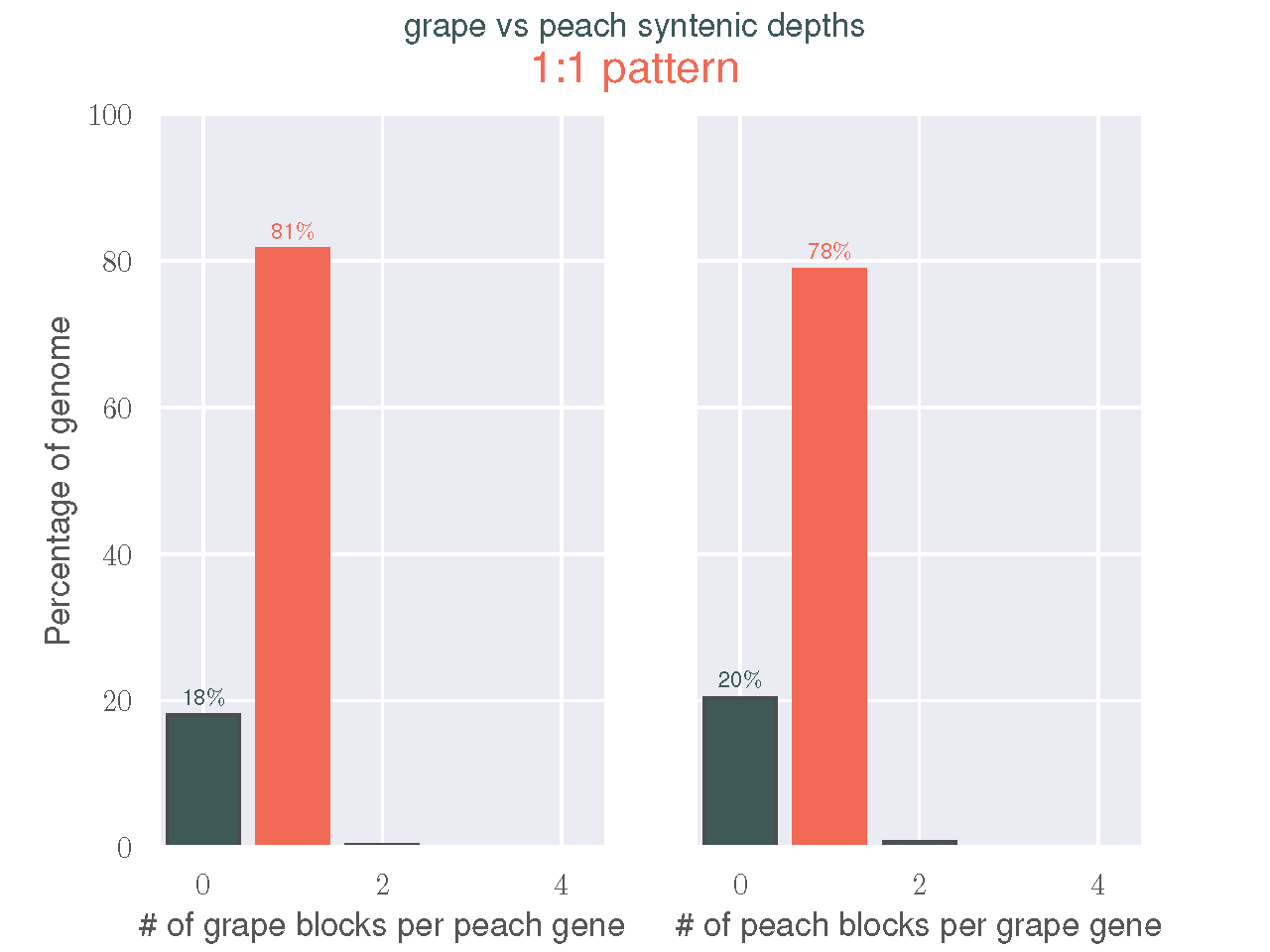
还可以手动查看synteny depth分布
python -m jcvi.compara.synteny depth --histogram grape.peach.anchors
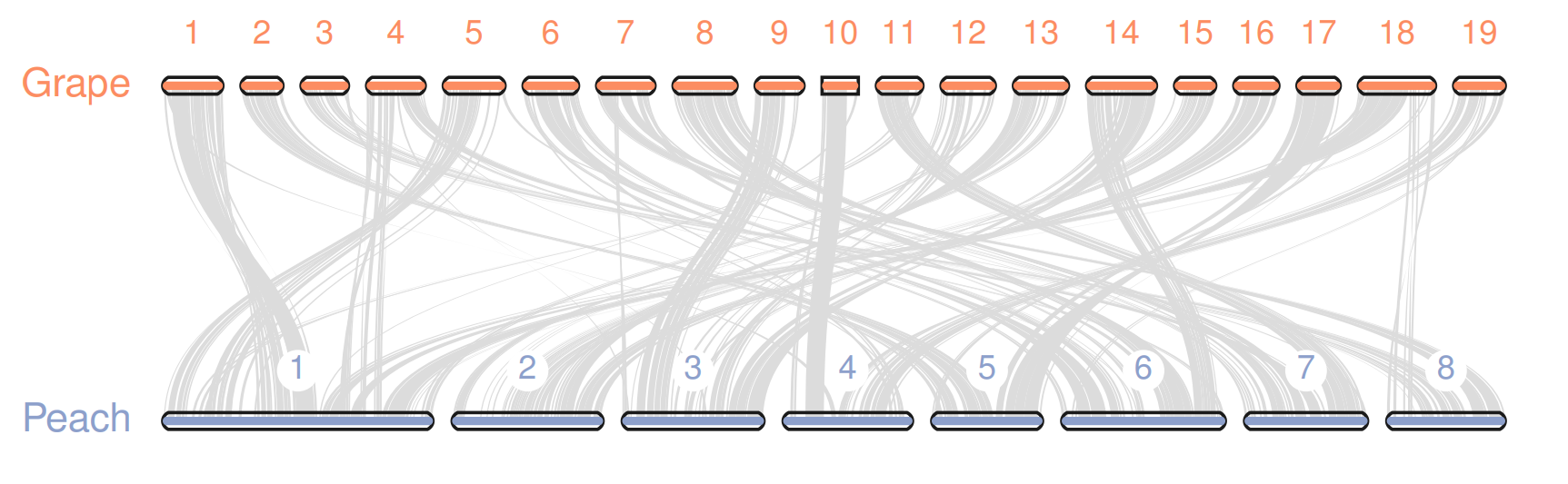
共线性图
首先先从.anchors文件中产生更简要的.simple文件
python -m jcvi.compara.synteny screen --minspan=30 --simple grape.peach.anchors grape.peach.anchors.simple
此外,除了原始BED文件和.simple文件文件,还需要手动设置两个文件用于绘图(注意,文件末尾不能存在多余的空行)
seqids:设定绘制哪些染色体,一般会去掉较小和未定位的scaffolds,下方设定了19个🍇的染色体与8个🍑的染色体chr1,chr2,chr3,chr4,chr5,chr6,chr7,chr8,chr9,chr10,chr11,chr12,chr13,chr14,chr15,chr16,chr17,chr18,chr19 Pp1,Pp2,Pp3,Pp4,Pp5,Pp6,Pp7,Pp8layout:设定画板上如何绘制。整体上canvas绘图的x,y轴的定位在0-1之间,前三行设定了染色体轨道的位置,循环,颜色,标签,垂直排列以及对应的BED文件。下方的track0就是🍇,track1就是🍑。下一节就是设定在哪些tracks之间绘制共线性关系。e,0,1代表着绘制track0与track1之间的共线性,使用grape.peach.anchors.simple文件中的详细信息。# y, xstart, xend, rotation, color, label, va, bed .6, .1, .8, 0, , Grape, top, grape.bed .4, .1, .8, 0, , Peach, top, peach.bed # edges e, 0, 1, grape.peach.anchors.simple
绘图
python -m jcvi.graphics.karyotype seqids layout
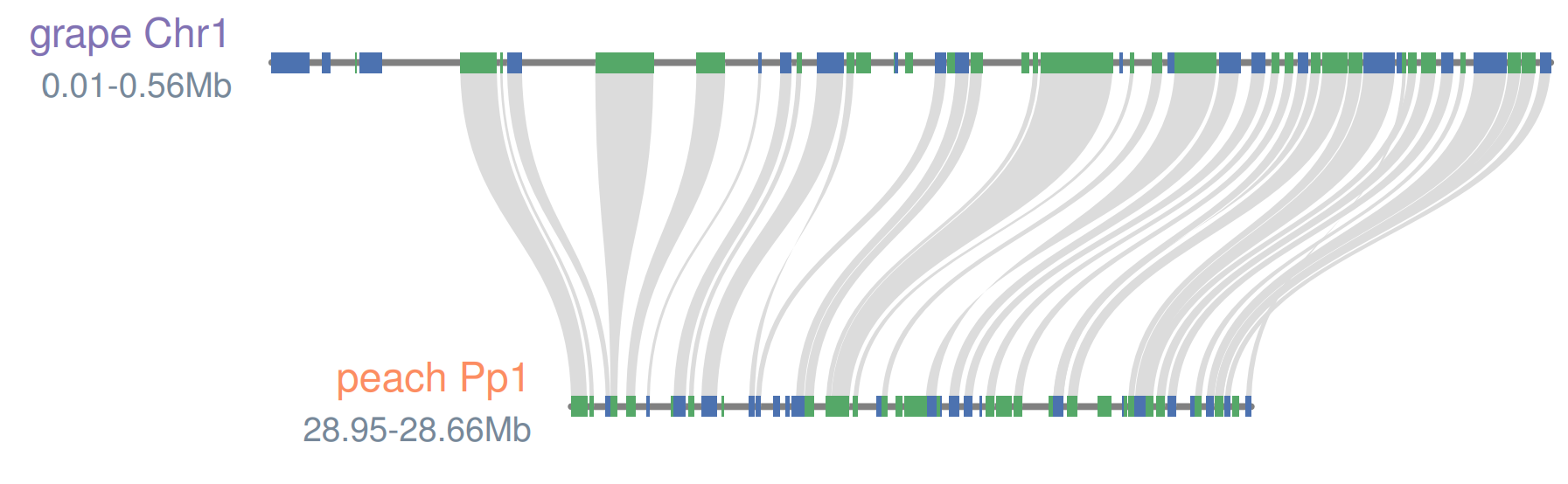
局部可视化
首先从基因水平提取匹配的区块
python -m jcvi.compara.synteny mcscan grape.bed grape.peach.lifted.anchors --iter=1 -o grape.peach.i1.blocks
参数--iter=1表明从每个🍇区域中提取1个最佳匹配区域。若将--iter设置为2,那么每个🍇区域就会有2个对应的🍑区域。这一点对于基因组重复区域的绘制很有帮助。
目前grape.peach.i1.blocks中包含有很多局部区域,我们可以手动选择需要绘制的部分。
head -50 grape.peach.i1.blocks > blocks.select
最终,我们仍然需要一个layout文件进行绘图,blocks.layout文件如下
# x, y, rot, ha, va, color, ratio, label
0.5, 0.6, 0, left, center, m, 1, grape Chr1
0.5, 0.4, 0, left, center, #fc8d62, 1, peach Pp1
# edges
e, 0, 1
然后,我们就可以进行绘制了
cat grape.bed peach.bed > grape_peach.bed
python -m jcvi.graphics.synteny blocks.select grape_peach.bed blocks.layout
调整与美化
设置颜色
species1.species2.anchors.simple
g*GSVIVT01012028001 GSVIVT01000604001 ppa011886m ppa008534m 392 +
GSVIVT01010441001 GSVIVT01000970001 ppa022891m ppa001358m 115 -
...
# g: green; r: red; etc.
多物种
layout
# y, xstart, xend, rotation, color, label, va, bed
.7, .2, .4, 45, , Grape, top, grape.bed
.5, .2, .8, 0, , Peach, top, peach.bed
.7, .4, .8, -45, , Cacao, bottom, cacao.bed
# edges
e, 0, 1, grape.peach.anchors.simple
e, 1, 2, peach.cacao.anchors.simple
seqids
chr1,chr2,chr3,chr4,chr5,chr6,chr7,chr8,chr9,chr10,chr11,chr12,chr13,chr14,chr15,chr16,chr17,chr18,chr19
scaffold_1,scaffold_2,scaffold_3,scaffold_4,scaffold_5,scaffold_6,scaffold_7,scaffold_8
scaffold_1,scaffold_2,scaffold_3,scaffold_4,scaffold_5,scaffold_6,scaffold_7,scaffold_8,scaffold_9,scaffold_10
三角形layout
# y, xstart, xend, rotation, color, label, va, bed
.5, .025, .625, 60, , Grape, top, grape.bed
.2, .2, .8, 0, , Peach, top, peach.bed
.5, .375, .975, -60, , Cacao, top, cacao.bed
# edges
e, 0, 1, grape.peach.anchors.simple
e, 1, 2, peach.cacao.anchors.simple
e, 0, 2, grape.cacao.anchors.simple