liuyujie0136's Website

A website for self learning, collecting and sharing.
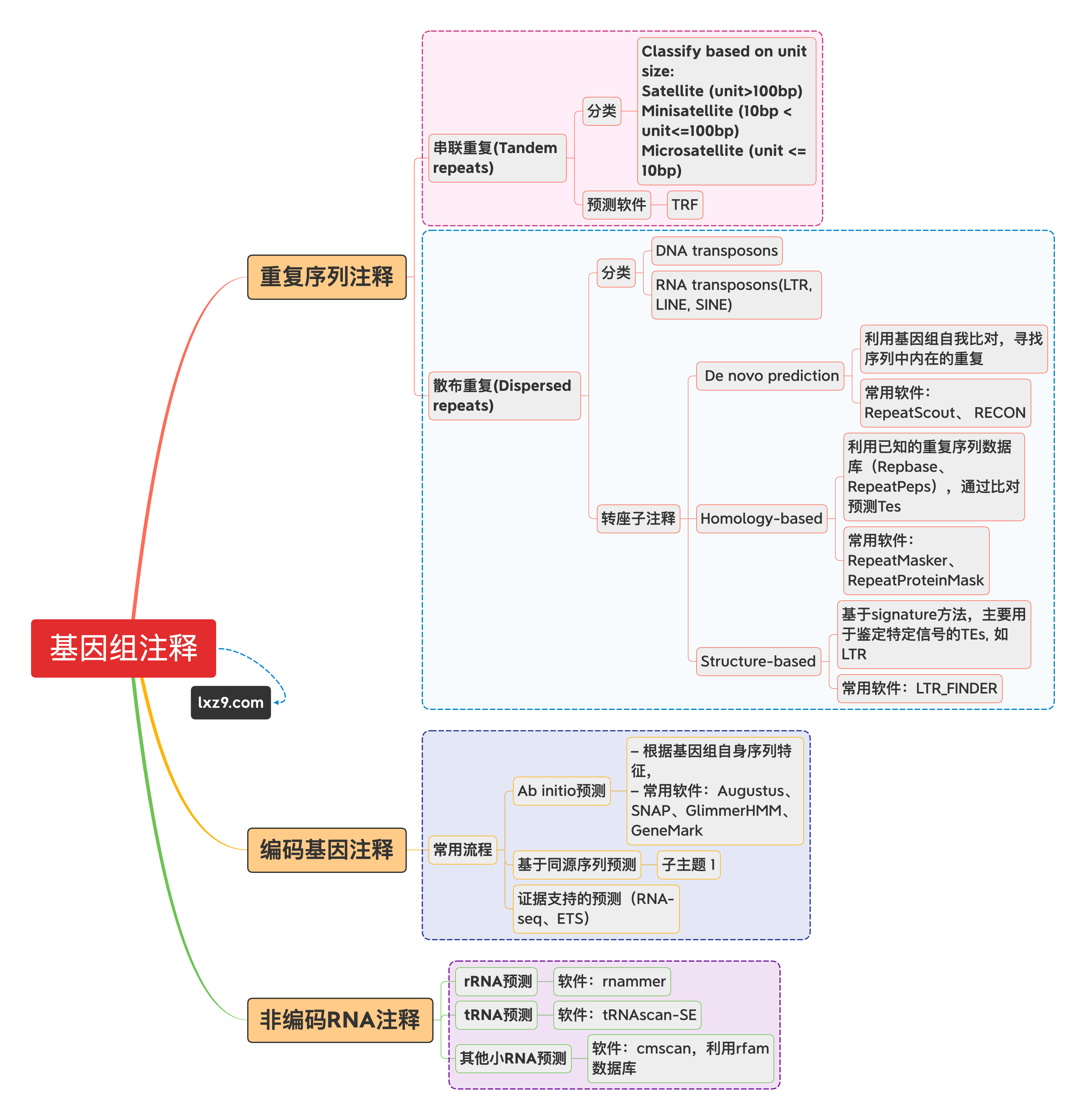
基因组注释基本流程
https://lxz9.com/2021/02/08/Annoation/
定义
基因组注释:是利用生物信息学方法和工具, 对基因组所有基因的生物学功能进行高通量注释, 是当前功能基因组学研究的一个热点。
基因组注释:即在一条DNA序列上, 通过从头、同源、结构定义等多种方法, 搜寻并定义基因组原件, 得到其位置、序列、结构、功能等信息。
基因组注释流程图
基因组注释前期准备
物种拉丁名, 例如:Orazy sativa , 基因ID:Osa000001
同源物种: 一般选5个左右物种, 需要有注释的基因/蛋白序列, 保证高组装和注释质量
转库组数据: RNAseq和lsoseq注释(用于结构注释中的转录辅助注释)(建议自测同样本的数据)
基因组注释的分析内容
重复注释
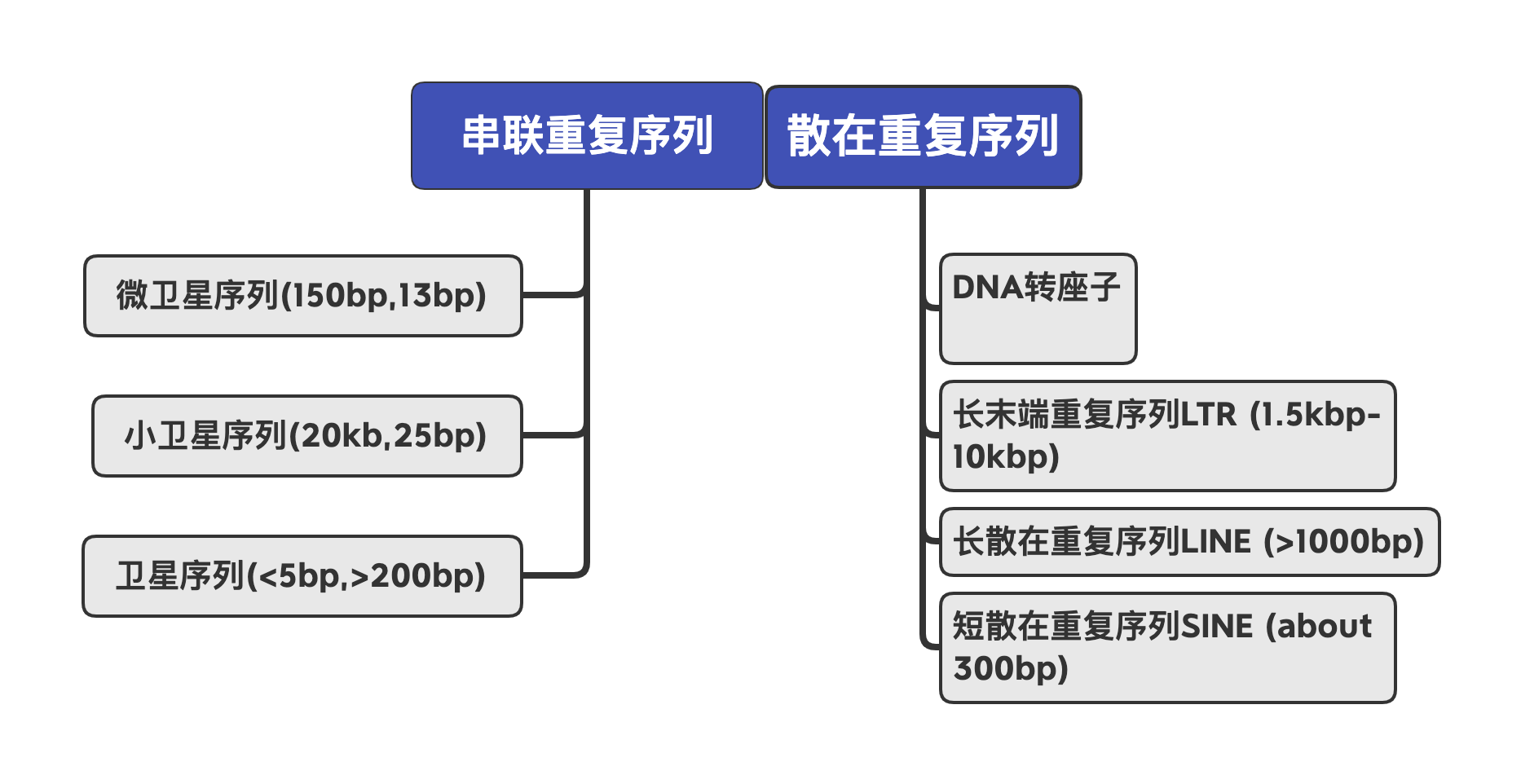
重复序列广泛存在于真核生物基因组中, 这些重复序列或集中成簇, 或分散在基因之间。根据分布把重复序列分为散在重复序列和串联重复序列。
重复序列根据序列特征分为2类:串联重复(Tandem repeats)和散布重复(Dispersed repeats)
RepeatMasker基于Repbase(DNA)/自建Elibrary查询重复序列
RepeatMasker -nolow -no_is -norna -parallel 2 -lib RepeatMasker.lib genome.fa
#-nohow:屏蔽低复杂简单重复; -no_is:跳过细菌插入元件检查; -norna:不掩盖小RNA(伪)基因;
#-parallel 并行使用的处理器数,可提升分析速度
RepeatProteinMask基于 Repbase(pep)查询重复序列
RepeatProteinMask -noLowSimple -pvalue 0.0001 genome.fa
#noLowSimple:关闭低复杂度和简单重复的屏蔽/注释; -pvalue:接受匹配的阈值
#注意点: genome.fa的D不能长于18个字符
TRF基于元件的结构特征等来识别重复序列
trf genome.fa 2 7 7 80 10 50 2000 -d -h
LTR-FINDER基于重复序列特征
Itr_finder -W 2 -C -s tRNAs.fa genome.fa
#-w 2 输出格式,2-table; -C:检测中心粒,删除高重复区域
repeatmodeler基于自身序列比对
BuildDatabase -name mydb genome.fa
RepeatModeler -database mydb -pa 6 >run.out
#-name:创建 database的名称;
#-pa:共享内存处理器的数量程序,可提升分析速度
结构注释
结构注释:注释可以产生具有生物学功能的蛋白的基因。一般包括启动子, 转录起始, 5’UTR, 起始密码子, 外显子, 内含子, 终止密码子, 3’UTR, poly-A等结构。
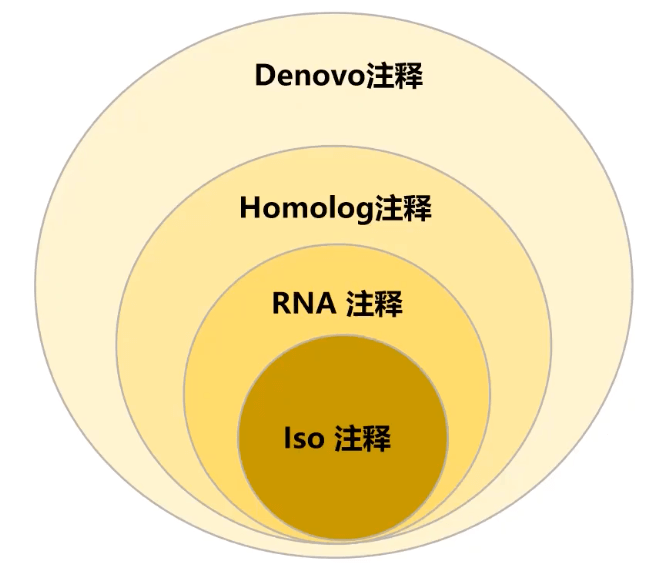
de novo 预测(屏蔽重复序列)
Augustus真核
augustus --species=XXX --AUGUSTUS CONFIG PATH= config --uniqueGeneld=true --nolnFrameStop=true--gff3=on --strand=both genome.mask.fa> genome.mask.fa.out
# --uniqueGeneld=true:gene:命名 aseqname.gn;
# --nolnFrameStop=true:不带有终止密码子的转录本;
# --gff3=on:输出格式gff3
GlimmerHMM真核,预测的基因数目较多长度较短,一般用于植物
glimmerhmm.genome.mask.fa -d XXX- f -g genome.mask.fa.gff
# -d 库de路径;
# -f:不要partial gene predictions;
# -g输出格式gff
Genscan真核,其预测的内含子较大,一般用于动物
genscan Humanlso.smat genome.mask.fa > genome.mask.fa.genscan
# Humanlsc.smat:参数文件,软件自带
4.其他软件
SNAP. GenelD GenemarkS
devnovo的软件很多,两个软件就可以了,太多软件会增加较多的假阳性,一般在 Augustus, GlimmerHMM, Genscan中选择即可
Homolog注释
利用近缘物种已知基因进行序列比对,找到同源序列。然后在同源序列的基础上,根据基因信号如剪切信号、基因起始和终止密码子对基因结构进行预测。
相对于从头预测的“大海捞针”,同源预测相当于先用一块磁铁在基因组大海中缩小了可能区域,然后从可能区域中鉴定基因结构。
利用TBlastn将同源物种的蛋白比对回基因组,得到候选区域。
利用 EXonerate/ Genewise进行精确的蛋白-核酸比对,以得到剪接位点。
Exonerate解决了 GeneWisez存在的很多问题,并且速度快了1000倍,默认选择EXonerate分析
RNA-seq辅助注释
- 将RNAseq数据进行
tophat比对; - 比对后的结果文件利用
cufflink构建转录本 - 使用
TransDecoder在构建的转录本上预测Open Reading Frame(ORF)。
Iso seq 辅助注释
- 将物种的三代全长转录本用
CD-HIT进行去冗余; - 将去冗余后的序列使用
gmap比对回基因组得到转录本位置; - 使用
TransDecoder在构建的转录本上预测 Open Reading Frame(ORF).
MAKERE整合
在基因组注释上, MAKER算是一个很强大的分析流程, 主要是进行 de novo 注释, Homolog注释, 转录辅助注释三者的整合, 保证最终注释基因集的可靠性
maker maker_exe.ctl maker_opts.ctl maker_bopts.ctl
#maker exe.ct:执行程序的路径
#maker_ boots.ctl: BLAST7和 Exonerate的过滤参数
#maker opts.ctl:其他信息,例如输入基因组文件,主要调整输入文件等( genome= ;est= ;protein= ;pred_gff= ;)
ncRNA注释
- rRNA(核糖体RNA)
与蛋白质结合形成核糖体,其功能是作为mn的支架,提供mRNA翻译成蛋白质的场所。 - tRNA(转运RNA)
·携带氨基酸进入核糖体,使之在mRNA指导下合成蛋白质。 - miRNA(miRNA)
·将mRNA降解或抑制其翻译,具有沉默基因的功能。 - SnRNA(小核RNA)
·主要参与RNA前体的加工过程,是RNA剪切体的主要成分。
miRNA与snRNA注释
- 采用Rfam和INFERNAL进行二级结构检测。
- ftp://ftp.sanger.ac.uk/pub/databases/Rfam
- blastn+cmsearch (INFERNAL程序)
rRNA注释
- 由于rRNA的结构保守程度非常高, 因此采用与已有的全长rRNA进行blastn比对而获得。
- blastn
tRNA注释
- 结构特点:三叶草型二级结构。
- 预测方法:针对二级结构进行检测。使用tRNAscan-SE
功能注释
功能注释:基因功能的注释依赖于上一步的基因结构预测, 根据预测结果从基因组上提取翻译后的蛋白序列和主流的数据库进行blastp比对, 完成功能注释。
常用数据库一共有以下几种:NR, KEGG, Uniprot (Swiss-Prot, TrEMBL), InterPro,Go
- KEGG
- 生物学通路数据库(Gene,Pathway,Ligand).
- http://www.genome.jp/kegg/
- blastp
- SWISS-PROT和TrEMBL
- UniProt (Universal Protein Resource)蛋白质序列数据库PIR、SWISS-PROT和TrEMBL统一起来, 建立了一个蛋白质数据库。
- http://www.uniprot.org/
- blastp
- Interpro
- 蛋白家族(protein families)、功能保守区域(domains)和功能位点(funtional sites)的数据库.
- http://www.ebi.ac.uk/interpro/
- InterProScan
- GO
- 基因功能注释数据库(GeneOntology)
- 三个层面Cellular Component、 Biological Process、 Molecular Function.
- http://www.geneontology.org/
- InterProScan
基因组评估 - BUSCO评估
BUSCO是一款使用python语言编写的对转录组和基因组组装质量进行评估的软件。在相近的物种之间总有一些保守的序列, 而BUSCO就是使用这些保守序列与组装的结果进行比对, 鉴定组装的结果是否包含这些序列, 包含单条、多条还是部分或者不包含等等情况来给出结果。
BUSCO软件根据OrthoDB数据库, 构建了几个大的进化分支的单拷贝基因集。将其与该基因集进行比较, 根据比对上的比例、完整性, 来评价准确性和完整性。
总结
基因组注释
- 重复注释: RepeatMask, RepeatProteinMask, TRF, LTR-FINDER,repeatmodeler
- 结构注释: Denovo注释, 同源注释, 转录辅助注释
- ncRNA注释: tRNA, rRNA, miRNA, snRNA
- 功能注释: NR, KEGG, InterPro, SWISS-PROT, TrEMBL, GO
- 基因组评估: BUSCO